近日,js555888金沙老品牌玉米生物学与遗传育种团队联合丹麦奥胡斯大学数量遗传中心等国家国际单位在《Genome Research》发表题为“Predicted protein 3D structure provides essential insights into the genetic architecture underlying phenotypic diversity in maize”的研究论文。
蛋白质是生命活动的重要承担者。蛋白质三维结构变异是蛋白质变异的重要体现,然而尚未有研究从群体水平上理解蛋白质三维结构变异对于表型多样性的贡献。为了探究蛋白质三维结构与表型之间的关系,本研究利用预测的蛋白质三维结构信息结合数量遗传学方法揭示了蛋白质三维结构对于表型多样性的贡献。
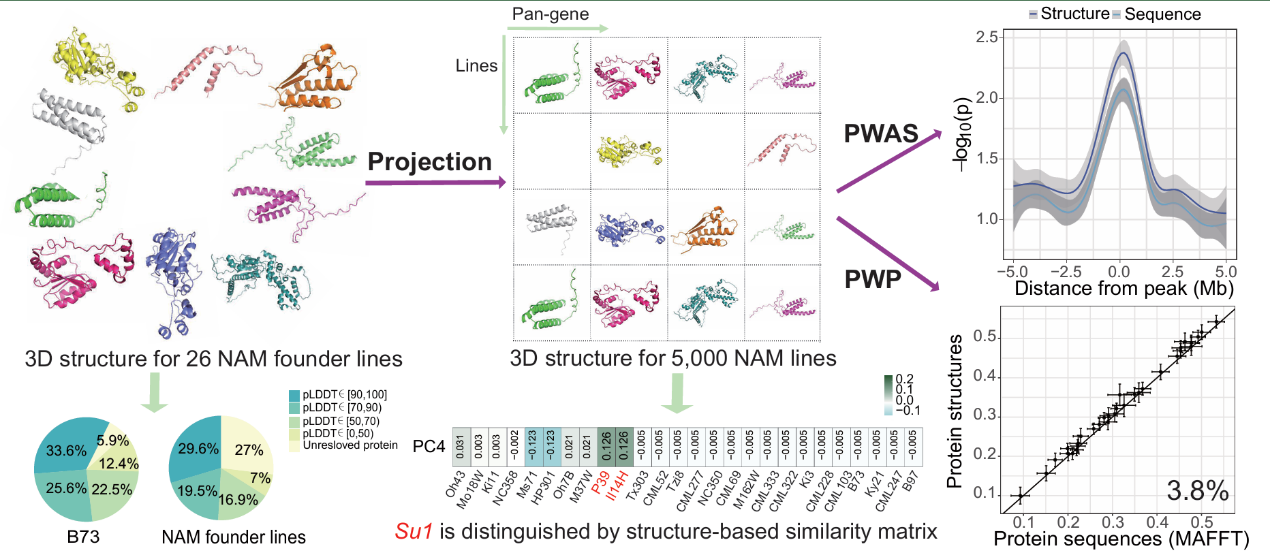
使用AlphaFold2预测了26个玉米NAM亲本自交系中795,649个蛋白质的三维结构。通过残基,蛋白及结构域三个层次说明了预测的蛋白质三维结构具有很高的置信度,可以用于后续群体遗传学和数量遗传学分析。将蛋白质结构注释到玉米Goodman自然群体,非同义突变/同义突变比例及衍生等位基因频率,GERP值,piN/piS等群体遗传学参数结果表明蛋白质结构中隐藏区域比暴露区域更容易受净化选择。
利用玉NAM群体的单倍型蛋白序列信息将亲本的蛋白质三维结构进行映射最终获得了约5000份的群体尺度的蛋白质结构信息(上图)。分别以蛋白质序列,蛋白结构作为基因型与表型进行关联分析(PWAS),结果表明蛋白质结构为基础的关联分析整体上有更高的统计显著性,并能鉴定到基于蛋白质序列关联所鉴定不到的候选基因。此外与传统的以SNP基础的GWAS相比,也能鉴定到一些独有的候选基因。
通过方差分解分析表明蛋白质结构层次的变异可以解释不同比例的表型变异。似然比检验表明加入蛋白质结构信息是表型变异的重要来源。使用32个已发表的表型性状进行全基因组预测(PWP),使用以蛋白结构为基础的全基因组预测比以序列为基础的全基因组预测平均提升了3.8%的表型预测准确性,使用以序列为基础的全基因组预测整合进蛋白结构变异信息比以序列为基础的全基因组预测平均提升了4.1%的表型预测准确性。
结果表明蛋白结构能检测到新的候选基因并能提升全基因组预测的准确性,对于新基因挖掘和全基因选择育种有潜在的促进作用。
js555888金沙老品牌硕士二年级研究生王帅为文章唯一第一作者。该研究由js555888金沙老品牌玉米团队主导,北京大学和丹麦奥胡斯大学为共同通讯单位,联合了美国康奈尔大学、美国农业部、美国德克萨斯先进计算中心等多家单位协同攻关。
论文链接:https://doi.org/10.1101/gr.280514.125
编辑:郭超
终审:吴清华
版权所有 js555888金沙(Macau)老品牌-Latest Official Website 我们的位置 您好,您是第位访客